모바일은 화면을 돌려 가로화면으로 보시는 게 읽으시기 편할 수 있습니다. 돌려서 보시는 걸 추천드릴게요!!

🩸 저번 업로드 내용에서 데이터를 불러오고, 앞으로 프로젝트를 진행할 방향을 알아보았습니다. 이번 포스팅에서는 데이터를 전처리해서 사용할 만한 데이터로 만드는 과정을 다룰 생각입니다. 먼저 데이터가 어떻게 생겼었는지 한번 더 살펴봅시다!!

🫀 Data Info
🩸 데이터는 위와 같이 생겼습니다. 이 데이터가 어떤 정보들을 가지고 있는지 알아봅시다.
cardio.info()>>
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 70000 entries, 0 to 69999
Data columns (total 13 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 id 70000 non-null int64
1 age 70000 non-null int64
2 gender 70000 non-null int64
3 height 70000 non-null int64
4 weight 70000 non-null float64
5 ap_hi 70000 non-null int64
6 ap_lo 70000 non-null int64
7 cholesterol 70000 non-null int64
8 gluc 70000 non-null int64
9 smoke 70000 non-null int64
10 alco 70000 non-null int64
11 active 70000 non-null int64
12 cardio 70000 non-null int64
dtypes: float64(1), int64(12)
memory usage: 6.9 MB
위에서 봤듯이 데이터는 70000명에 대한 정보를 담고 있습니다. 또한 weight 를 제외한 각각의 attribute는 int64 의 자료형을 가지고 있으며, 다행히 결측값은 하나도 없는 것을 알 수 있었습니다.
🫀 Data adjustment
🩸 위의 데이터에서 weight와 height attribute 가 있는 이유는 각 피험자들의 비만율을 알아보기 위해서라고 생각했기 때문에, 이를 좀 더 보편적인 수치로 바꿔주기 위해서 BMI 라는 수치를 사용하기로 했습니다. 또한 위의 데이터에서 age 가 상당히 큰 수임을 확인했습니다. 아마 살아온 날짜로 나이를 표현한 것 같아, 각각을 365로 나누고 소수점 첫째자리에서 반올림해 일반적인 연령으로 나이를 바꿔주었습니다.
🩸 그리고 나서 사용하지 않을 attribute인 id, height, weight를 모두 삭제했습니다. 전처리를 위한 코드는 아래와 같습니다.
cardio['age'] = cardio['age'] / 365
cardio['age'] = round(cardio['age'], 0).astype('int64').copy()
cardio['height'] = cardio['height'] / 100
cardio['BMI'] = cardio['weight'] / (cardio['height']**2)
cardio['BMI'] = round(cardio['BMI'], 2).copy()
cardio = cardio.drop(['id','height','weight'], axis = 1)
cardio.head()
🫀 Blood pressure preprocessing
저희가 생각하기에 심혈관 질환에 가장 큰 영향을 미치는 것은 혈압이라고 생각했기 때문에, 데이터에서 혈압에 대한 부분은 한번 짚어봐야겠다고 생각했습니다. 따라서 ap_hi(수축기혈압) 와 ap_lo(이완기혈압) attribute 각각의 info를 확인했습니다.
pd.DataFrame({'ap_hi' : cardio['ap_hi'].describe(), 'ap_lo' : cardio['ap_lo'].describe()})>>
ap_hi ap_lo
count 70000.000000 70000.000000
mean 128.817286 96.630414
std 154.011419 188.472530
min -150.000000 -70.000000
25% 120.000000 80.000000
50% 120.000000 80.000000
75% 140.000000 90.000000
max 16020.000000 11000.000000
분명히 혈압에 대한 attribute 임에도 음수 또는 max 값이 10,000 이 넘는 경우가 있음을 확인할 수 있었습니다. 이러한 값들이 얼마나 퍼져있는지 알아보기 위해서 ap_hi, ap_lo 에 대한 box plot을 그려보았습니다.
# plotly library를 사용해서 Box plot 생성
fig = go.Figure()
fig.add_trace(go.Box(y=cardio['ap_hi'], name = 'ap_hi'))
fig.add_trace(go.Box(y=cardio['ap_lo'], name = 'ap_lo'))
fig.show()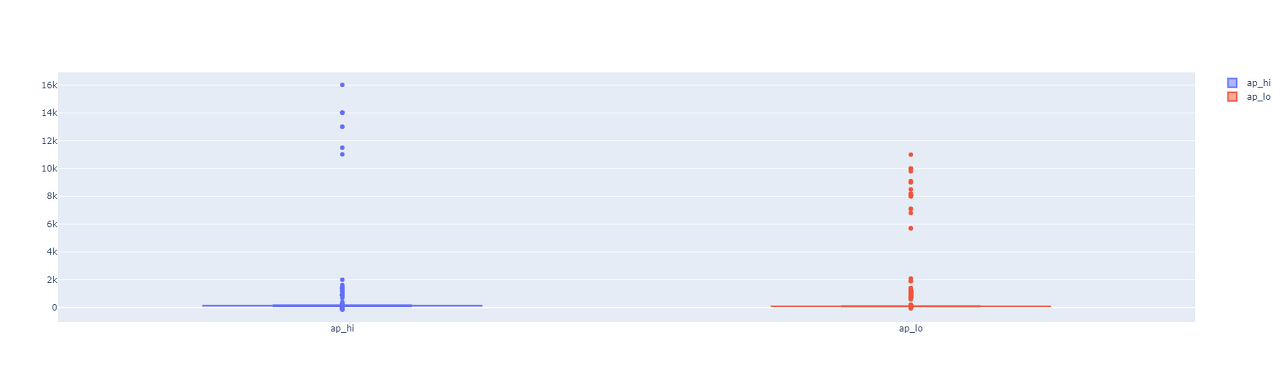
본 글에 있는 box plot은 출력된 그래프를 캡처한 것이라 보이지 않지만, iplot을 사용해서 그래프를 그리면 사용자의 마우스 커서에 반응하여 각 box plot의 upper fence / lower fence를 알 수 있습니다. 이때 각각을 정리하면 아래와 같습니다.
▪ ap_hi : upper_fence = 170 / lower_fence = 90
▪ ap_lo : upper_fence = 105 / lower_fence = 65
이제 이 값들의 이상 / 이하에 속하는 값들을 outlier로 생각해서 없애주는 방법으로 전처리할 것입니다. 또한, 일반적으로 혈압이 수축할 때가 이완할 때 보다 높아야 할 것이기에 ap_hi가 ap_lo보다 낮은 행 역시 삭제해 줄 생각입니다.
# ap_hi (수축혈압)가 ap_lo (이완혈압)보다 낮은 행 삭제
# 조건을 만족하는 row의 index를 받아서 삭제할 인덱스로 저장 - Index 자료형
# .drop() 함수를 사용해서 삭제 진행 - 저장한 인덱스를 parameter로 받아 row 삭제
low_drop_index = cardio[(cardio['ap_hi'] < cardio['ap_lo'])].index
cardio = cardio.drop(low_drop_index).copy()
# Ap_hi Preprocessing
# 전자와 동일
drop_index_sys = cardio[(cardio['ap_hi'] < 90) | (cardio['ap_hi'] > 170)].index
cardio = cardio.drop(drop_index_sys).copy()
# Ap_lo Preprocessing
# 전자와 동일
drop_index_dias = cardio[(cardio['ap_lo'] < 65) | (cardio['ap_lo'] > 105)].index
cardio = cardio.drop(drop_index_dias).copy()
len(cardio)>> 64500
이렇게 혈압에 대한 전처리 결과 70000개의 행 중에서 64500개의 행만 남은 것을 확인할 수 있습니다. 전처리된 데이터에 대한 box plot을 그려 전반적인 분포를 다시 확인해보겠습니다.
# plotly library를 사용해서 Box plot 생성
fig = go.Figure()
fig.add_trace(go.Box(y=cardio['ap_hi'], name = 'ap_hi'))
fig.add_trace(go.Box(y=cardio['ap_lo'], name = 'ap_lo'))
fig.show()
🩸 이렇게 해서 주요 attribute의 전처리를 해보았습니다. 다음 글에서는 다른 attribute의 경우에는 이런 noisy data나 case에 해당하지 않는 경우가 있는지 확인해보겠습니다.
'🐍 파이썬 데이터 분석 프로젝트 > 🫀 심혈관질환 데이터 분석' 카테고리의 다른 글
| 🫀 심혈관질환 데이터 분석 06. 패턴분석 데이터 전처리 (0) | 2023.01.31 |
|---|---|
| 🫀 심혈관질환 데이터 분석 05. 상관관계 분석 (0) | 2023.01.31 |
| 🫀 심혈관질환 데이터 분석 04. attribute 노이즈 확인 (0) | 2023.01.29 |
| 🫀 심혈관질환 데이터 분석 02. 데이터 소개 (0) | 2023.01.29 |
| 🫀 심혈관질환 데이터 분석 01. readme (0) | 2023.01.29 |