모바일은 화면을 돌려 가로화면으로 보시는 게 읽으시기 편할 수 있습니다. 돌려서 보시는 걸 추천드릴게요!!

🩸 저번 글에서는 우리가 연관관계 분석을 통해 얻은 attribute set 5와 전처리만을 수행한 데이터, PCA 결과 만들어진 주성분 3가지 경우에 대해 결정 트리 모델을 만들었습니다. 이번 글에서는 결정 트리들이 모여 만들어지는 랜덤 포레스트를 통해 모델의 예측력을 시험해보도록 합시다.
🩸 만든 랜덤 포레스트에 대해 ROC Curve와 Confusion Matrix를 만들어 각 모델의 성능이 어떤지 비교해볼 생각입니다.
🫀 1. Attribute Set 5 : age, aphi, aplo, cholesterol, gluc, BMI, active 랜덤포레스트
📌 사용할 라이브러리는 아래와 같습니다.
# 데이터 셋을 train / test 로 분류해주는 라이브러리
from sklearn.model_selection import train_test_split
# 랜덤포레스트를 위한 데이터 정규화 - StandardScaler 라이브러리
# 랜덤포레스트 생성 RandomForestClassifier 라이브러리
# acuuracy_score 라이브러리 : 정확도 계산 함수
from sklearn.preprocessing import StandardScaler
from sklearn.ensemble import RandomForestClassifier
from sklearn.ensemble import GradientBoostingClassifier
from sklearn.metrics import accuracy_score
# ROC Curve, Confusion Matrix, classification report 생성을 위한 라이브러리
from sklearn.metrics import roc_curve, auc
from sklearn.metrics import (
classification_report, confusion_matrix,
ConfusionMatrixDisplay)
from enum import Enum
랜덤 포레스트 구현 방법은 결정 트리와 크게 다르지 않습니다. 데이터를 train set과 test set으로 나누고, 랜덤 포레스트 모듈을 사용하면 됩니다. 저희가 정해줄 것은 포레스트에 위치할 각 트리들의 최대 깊이인데, 12일때 그 결과가 가장 좋게 나왔기에 max_depth를 12로 설정했습니다.
# attribute set 5를 사용한 경우
cardio.target_out = cardio['cardio'].copy()
cardio.feat_out = cardio.drop(['cardio','gender','alco','smoke'], axis = 1).copy()
train_x_out, test_x_out, train_y_out, test_y_out =
train_test_split(cardio.feat_out, cardio.target_out, test_size=0.3, random_state=0)
# 데이터 표준화 작업
sc = StandardScaler()
sc.fit(train_x_out)
# 표준화된 데이터셋
train_x_out_std = sc.transform(train_x_out)
test_x_out_std = sc.transform(test_x_out)
clf = RandomForestClassifier(random_state=0, max_depth = 12, n_estimators = 200)
clf.fit(train_x_out,train_y_out)
predict6 = clf.predict(train_x_out)
predict5 = clf.predict(test_x_out)
print("Train set accuracy : ", accuracy_score(train_y_out, predict6))
print("Test set accuracy : ", accuracy_score(test_y_out,predict5))>>
Train set accuracy : 0.7655592469545958
Test set accuracy : 0.7276485788113695
🫀 2. Original Data Random Forest
# original data의 모든 attribute를 사용한 경우 : case 1
cardio.target_all = cardio['cardio'].copy()
cardio.feat_all = cardio.drop(['cardio'], axis = 1).copy()
train_x_all, test_x_all, train_y_all, test_y_all =
train_test_split(cardio.feat_all, cardio.target_all, test_size=0.3, random_state=0)
# 데이터 표준화 작업
sc = StandardScaler()
sc.fit(train_x_all)
# 표준화된 데이터셋
train_x_std_all = sc.transform(train_x_all)
test_x_std_all = sc.transform(test_x_all)
clf = RandomForestClassifier(random_state=0, max_depth = 12, n_estimators = 200)
clf.fit(train_x_all,train_y_all)
predict4 = clf.predict(train_x_all)
predict3 = clf.predict(test_x_all)
print("Train set accuracy : ", accuracy_score(train_y_all, predict4))
print("Test set accuracy : ", accuracy_score(test_y_all,predict3))>>
Train set accuracy : 0.7690586932447397
Test set accuracy : 0.7299741602067183
먼저 original data에 대해 만들어진 랜덤 포레스트를 바탕으로 ROC Curve와 Confusion Matrix를 시각화할 것입니다.
# ROC Curve
# 전체 attribute를 사용한 경우의 ROC curve 출력
fpr_tr, tpr_tr, ths_tr = roc_curve(test_y_all, predict3)
auc_tr = auc(fpr_tr, tpr_tr)
fig = plt.figure(figsize = (8,4))
plt.subplot(121)
plt.plot(fpr_tr, tpr_tr,
lw=3, label='ROC curve (area = %0.2f)' % auc_tr)
plt.plot([0, 1], [0, 1], color='skyblue', lw=2,linestyle='--')
plt.xlabel('fpr')
plt.ylabel('tpr')
plt.legend(loc="lower right", prop={'size' : 10})
plt.title('validation set')
plt.show()
# Confusion Matrix
# 전체 attribute를 사용한 경우의 confusion matrix 출력
class Diagnosis(Enum):
No_Cardio = 0
Cardio = 1
cm = confusion_matrix(
test_y_all,
(predict3>.5).astype(np.int8),
# normalize='true',
)
disp = ConfusionMatrixDisplay(
confusion_matrix=cm,
display_labels=[d.name for d in Diagnosis],
)
disp.plot(ax=plt.subplots(1, 1, facecolor='white')[1], cmap=plt.cm.Reds)

📌 original data에 대한 random forest의 결과를 해석하면 아래와 같습니다.
- 결정 트리를 사용한 경우에 비해 향상된 accuracy를 보여줍니다.
- 하지만 좋은 결과를 얻었다고 하기엔 어렵습니다
- 따라서 PCA를 통한 랜덤 포레스트도 수행해 볼 예정입니다.
🫀 3. PCA Dataframe Random Forest
PCA_df_target = PCA_df['cardio']
PCA_df_feat = PCA_df.drop(['cardio'],axis = 1)
train_x_PCA, test_x_PCA, train_y_PCA, test_y_PCA =
train_test_split(PCA_df_feat, PCA_df_target, test_size=0.3, random_state=1)
clf = RandomForestClassifier(random_state=1)
clf.fit(train_x_PCA,train_y_PCA)
predict2 = clf.predict(train_x_PCA)
predict1 = clf.predict(test_x_PCA)
print("Train set accuracy : ", accuracy_score(train_y_PCA, predict2))
print("Test set accuracy : ", accuracy_score(test_y_PCA,predict1))>>
Train set accuracy : 0.9999114064230343
Test set accuracy : 0.7422739018087855
🚩 ROC Curve와 Confusion Matrix를 출력하겠습니다.
# PCA DataFrame을 사용한 경우의 ROC curve 출력
fpr_tr, tpr_tr, ths_tr = roc_curve(test_y_PCA, predict1)
auc_tr = auc(fpr_tr, tpr_tr)
fig = plt.figure(figsize = (8,4))
plt.subplot(121)
plt.plot(fpr_tr, tpr_tr,
lw=3, label='ROC curve (area = %0.2f)' % auc_tr)
plt.plot([0, 1], [0, 1], color='skyblue', lw=2,linestyle='--')
plt.xlabel('fpr')
plt.ylabel('tpr')
plt.legend(loc="lower right", prop={'size' : 10})
plt.title('validation set')
plt.show()
# PCA DataFrame을 사용한 경우의 confusion matrix 출력
class Diagnosis(Enum):
No_Cardio = 0
Cardio = 1
cm = confusion_matrix(
test_y_PCA,
(predict1>.5).astype(np.int8),
# normalize='true',
)
disp = ConfusionMatrixDisplay(
confusion_matrix=cm,
display_labels=[d.name for d in Diagnosis],
)
disp.plot(ax=plt.subplots(1, 1, facecolor='white')[1], cmap=plt.cm.Blues)
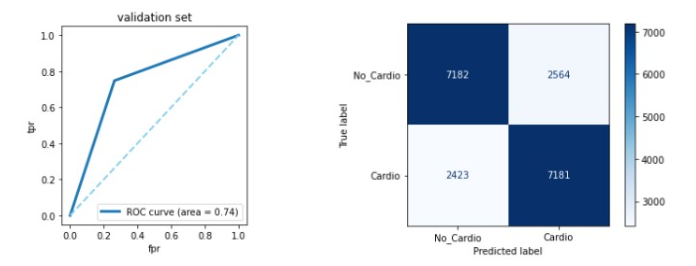
📌 PCA Dataframe을 사용해서 랜덤 포레스트를 돌린 결과는 아래와 같이 해석할 수 있습니다.
- 결정 트리를 사용한 결과에 비해 좋은 결과를 보여줍니다.
- ROC Curve와 Confusion Matrix를 보면 Original Data를 사용한 경우보다 나은 예측력을 보여줍니다.
- 하지만 train set의 accuracy와 test set의 accuracy 차이가 너무 크기 때문에 오버피팅이 일어났음을 알 수 있습니다.
💡 4. 프로젝트 결론
💡 데이터의 object가 많아 모델을 적용시켰을 때 accuracy가 좋을 것이라 예상했는데, 생각했던 것보다는 좋지 않았습니다.
💡 이러한 결과들 중에서 가장 나은 성능을 보여준 경우를 뽑으면 아래와 같습니다.
- 🏆 전체 attribute를 사용한 경우의 raandom forest 모델
- 🏆 attribute set 5 : age, ap_hi, ap_lo, cholesterol, gluc, active, BMI 를 사용한 경우의 random forest 모델
💡 원본 데이터 test set의 전체 attribute에 대한 Decision Tree의 accuracy가 max_depth=5 일때 0.72439로 가장 높았습니다. Accuracy가 생각보다 낮게 나왔기 때문에 오버피팅을 첫번째 염두에 두고 train set에 대한 accuracy를 산출해 보았습니다. 하지만 max_depth=5 일때 model의 train set에 대한 accuracy 역시 0.726을 조금 상회하는 수준이었고, 이로부터 오버피팅이라기 보다는 학습 자체가 제대로 되지 않았을 가능성이 높다고 생각하였습니다. 다만, 트리의 최대 깊이를 설정하지 않고 default 상태인 None으로 학습을 진행한 경우에는 test set에서 0.642 정도의 accuracy로 그 성능이 더욱 떨어진 것을 확인하였습니다. 이에 그 경우의 train set에 대한 accuracy를 구한 결과 대부분의 수치가 0.9를 상회하는 값이 나왔으며, 확실히 오버피팅이 발생한 것을 확인할 수 있었습니다. 이를 보아 Decision Tree 학습과 테스트에 있어서 Pruning이 중요하다는 것으로 해석할 수 있을 듯 합니다. 반면에, 2절의 연관관계 분석을 통해 추출한 attribute를 통한 accuracy는 전체 attribute에 의한 accuracy와 비슷한 수치를 보여주며 연관관계 분석의 유효함을 입증하였습니다. 또한, 이를 Tree들의 집합인 Random Forest에서 분류하였을 때는 max_depth=12 인 경우에 0.72997 로 소폭 상승했습니다. 여러 개의 트리들을 통해 classification을 한 결과로는 그렇게 좋아보이는 상승량은 아니지만, 성능이 조금이나마 향상되는 것을 확인할 수 있었습니다.
💡 다음으로는 PCA 분석을 통해 얻은 principal component들을 사용하여 classification을 진행하였습니다. 우선 Decision Tree의 경우에는 max_depth=6에서 가장 높은 accuracy를 가졌으며, 그때의 결과는 0.714 정도였습니다. 따라서 이 경우에도 오버피팅을 생각하고 train set에 대한 accuracy를 산출했으나, 그 경우의 accuracy 역시 0.72를 조금 상회하는 결과가 나왔습니다. 따라서 이 경우에도 학습이 제대로 이루어지지 않았다는 결론을 내릴 수 있었습니다. 또한 max_depth를 None로 설정했을 때에는 train set에 대한 accuracy가 0.99로 완전히 학습에 성공한 반면, test set accuracy는 0.71 정도로 오히려 성능이 살짝 떨어졌습니다. 이 경우 역시 오버피팅이 발생한 것으로 사료되며, pruning의 중요성을 입증한 경우라고 볼 수 있을 것 같습니다. 이어서 진행한 Random Forest에서는 test set에 대한 accuracy가 0.742 정도로 원래 데이터를 사용해서 Random Forest를 수행했을 때보다 높은 성능의 향상을 보여주었습니다. 하지만 이때의 train set에 대한 accuracy가 0.99가 나왔기 때문에 오버피팅이 일어났다고 결론을 내렸습니다.
💡 일반적으로 우리는 test set에 대한 accuracy가 높은 모델을 선호할 수 밖에 없습니다. 아무래도 데이터를 입력했을 때 가장 먼저 눈에 보이는 지표이고, 정답을 많이 예측하는 모델이 우리가 만들기를 원하는 모델이기 때문입니다. 이러한 통념 하에서는 이번 프로젝트에 있어 PCA 분석을 통한 Random Forest 모델이 0.742 정도의 accuracy로 가장 좋은 모델이라고 말할 것입니다. 하지만 이는 모델이 train set에 대해 오버피팅이 일어났다는 사실을 고려하지 않은 결론입니다. 물론 오버피팅이 있더라도 test set의 결과가 눈에 띄게 좋다면 그 모델을 고르는 것이 맞겠지만, 각각의 accuracy들은 분포가 거의 균일한 값들을 가지고 있습니다. 따라서, 바라던 만큼의 좋은 결과가 나오지는 않았으나 위에서 언급한 모델에서 하나를 고른다면 연관관계 분석을 통해 추출한 attribute set (5) 를 사용하여 Random Forest를 수행하였거나, 원본 데이터의 전체 attribute를 가지고 Random Forest를 수행한 모델을 선택하는 것이 최선의 선택이 될 것 같습니다. Accuracy가 그렇게 높게 나오지 않은 이유는 아무래도 object는 많지만 target의 분포가 balanced하고, dimension이 큰 데이터가 아니기 때문일 것입니다. 즉, target의 균일한 분포를 설명하기에는 dimension이 부족했다는 의미입니다. 따라서 굳이 dimensionality reduction을 하지 않아도 모델을 학습시키는 데 무리가 없는, 즉 원본 그 상태로도 정보가 부족했을 가능성이 있는 데이터이지 않을까 생각합니다.
🩸 이번 글을 끝으로 2022년도 1학기에 수행한 데이터마이닝 프로젝트 정리를 끝냈습니다. 프로젝트가 끝이 나고 한참 후에 올린 글들이기 때문에 군데군데 미흡함이 있을 수 있습니다만, 이렇게 정리하면서 프로젝트 진행 중에는 찾지 못한 몇가지 오류들을 발견할 수 있었고, 새롭게 공부하는 느낌이 들어 좋았습니다. 통계학 공부중이기도 하고 졸업 프로젝트 진행중이기도 해서 데이터마이닝 이론을 언제 정리할 수 있을지는 모르겠지만, 가급적이면 빨리 정리해보겠습니다😊😊.
🩸 아마 다음 글은 프로젝트 결과 레포트가 될 듯 합니다. 프레젠테이션 자료도 올릴 수 있다면 올리겠습니다!!
'🐍 파이썬 데이터 분석 프로젝트 > 🫀 심혈관질환 데이터 분석' 카테고리의 다른 글
| 🫀 심혈관질환 데이터 분석 16. 최종 분석 결과 레포트 (0) | 2023.02.04 |
|---|---|
| 🫀 심혈관질환 데이터 분석 14. 결정트리 시각화 (0) | 2023.02.03 |
| 🫀 심혈관질환 데이터 분석 13. 결정트리 구현 (0) | 2023.02.03 |
| 🫀 심혈관질환 데이터 분석 12. 라이브러리 임포트 (0) | 2023.02.03 |
| 🫀 심혈관질환 데이터 분석 11. plotly 차트보드 (0) | 2023.02.02 |